Ny metode gør det muligt at identificere proteinubalance bag sygdomme som kræft
Forskere fra Københavns Universitet har udviklet en spritny metode kaldet Deep Visual Proteomics. Metoden gør det muligt at kombinere cellers visuelle egenskaber med deres molekylære profil, hvilket kan give forskerne helt ny viden om sygdomme som kræft.
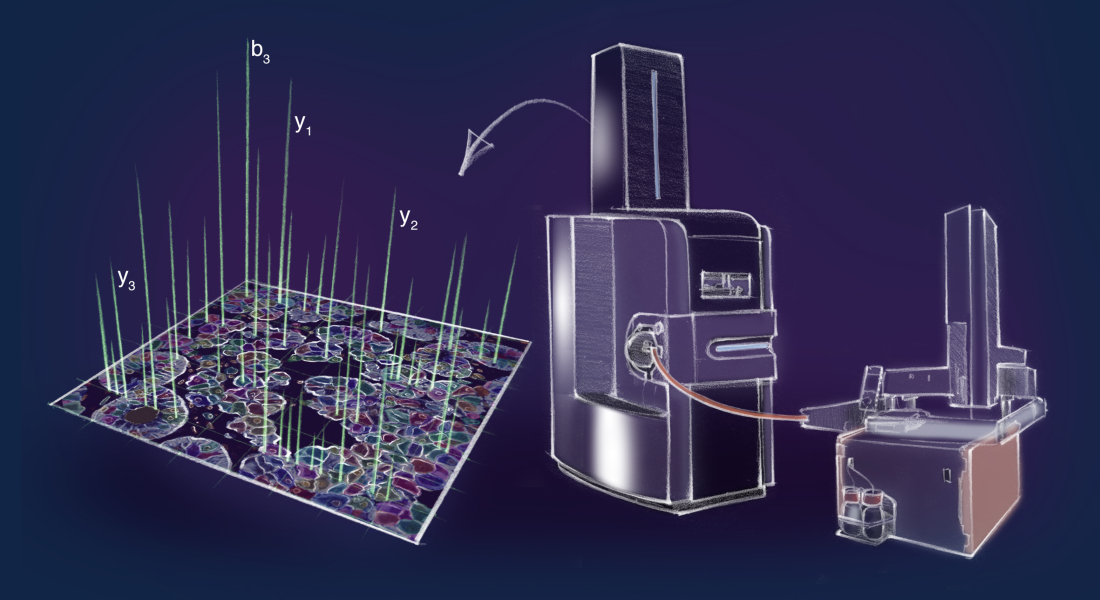
Når kroppen er syg, skyldes det en ubalance i de mindste af vores byggesten, proteinerne. Nu har forskere fra Det Sundhedsvidenskabelige Fakultet på Københavns Universitet og Max Planck Institute of Biochemistry udviklet en banebrydende metode kaldet Deep Visual Proteomics (DVP), som kan hjælpe dem til at forstå, præcis hvad der går galt i sygt væv, som eksempelvis kræfttumorer.
DVP kan blandt andet bruges til at undersøge, hvorfor kemoterapi ikke virker på en given patient, eller til at identificere den proteinubalance, som er årsag til sygdommen.
”Når kemoterapi ikke virker, er det, fordi nogle af tumorcellerne er kemoresistente. Ved at dissekere en kemoresistent tumor og sammenligne sygt væv med raskt væv fra samme patient, kan vi finde ud af, hvilke af tumorcellerne der er årsag til resistensen. Ved hjælp af DVP kan vi beskrive proteinlandskabet i syge celler og dermed identificere proteinubalancen. Det gør os i stand til at hjælpe lægerne med at vælge den helt rigtige behandling,” siger lektor Andreas Mund, Novo Nordisk Foundation Center for Protein Research, Det Sundhedsvidenskabelige Fakultet, Københavns Universitet.
Ifølge forskerne kan DVP også øge diagnose- og behandlingsmulighederne i forbindelse med andre sygdomme, herunder stofskiftesygdomme og neurodegenerative lidelser.
“Det er proteinerne, der får kroppen til at fungere – eller svigte. Sygdomme opstår, når der er ubalance i kroppens proteiner. Vores metode gør det ikke bare muligt at beskrive proteinlandskabet i disse celler i hidtil uset detaljeringsgrad, så vi kan se, præcis hvor problemet er. Den gør det også muligt at forbinde proteinprofilen med cellens visuelle kendetegn. Kunstig intelligens hjælper os med at opdage særligt informative, men ellers skjulte mønstre, der er for fine eller komplekse, til at vi kan skelne detaljerne uden hjælp. Disse proteinsignaturer kan hjælpe os til bedre at forstå mekanismerne bag den pågældende sygdom. Det er et helt nyt redskab, der kan hjælpe klinikere til at udvikle bedre diagnose- og behandlingsstrategier,” siger Andreas Mund.
Et proteinisbjerg
Viden om proteinlandskabet er værdifuld, fordi proteinerne er afgørende for vores sundheds- eller sygdomstilstand og udgør vigtige lægemiddelmål. Hvis du er syg og tager til lægen, er der sandsynlighed for, at hun vil tage en blodprøve. Når lægen derpå undersøger dit blod, vil hun lede efter uregelmæssigheder i bestemte proteiner for at finde ud af, om du har betændelse i kroppen.
Hvor lægen kigger på måske 10-20 proteiner, kan DVP analysere tusindvis af proteiner på én gang og identificere særlige fællestræk. På den måde kan klinikere trække en streg fra cellernes morfologiske træk, som er synlige i et mikroskop, til deres molekylære profil, hvilket giver betydeligt større indblik i, hvad der rent faktisk sker under celleoverfladen. Den viden kan vise sig at være en stor hjælp, når det kommer til at stille den rette diagnose eller finde frem til den bedste behandling.
“Man kan tænke på det som et isbjerg. Cellens morfologiske træk såsom formen udgør den synlige del af isbjerget. Men det meste af isbjerget er stadig skjult under overfladen. Det er cellens molekylære profil, proteinlandskabet. Man kan ikke behandle en sygdom ordentligt, hvis man kun kan se toppen af isbjerget. Vi har brug for det fulde billede,” forklarer Andreas Mund.
Deep Visual Proteomics: En yderst avanceret metode
For at vise, at metoden virker, har forskerne anvendt den på vævsprøver fra melanomer (hudkræft) og acinære cellekarcinomer (kræft i spytkirtlerne), som de har modtaget fra danske biobanker.
Ved hjælp af kunstig intelligens inddeler DVP automatisk tumorcellerne i undergrupper på basis af visuelle kendetegn såsom form og proteinplacering. Cellerne overføres derpå til et lasermikroskop til mikrodissektion, som skiller cellerne fra hinanden og placerer dem i brønde for de forskellige undergrupper. Ved hjælp af en spritny, ultrasensitiv proteomicsmaskine kan forskerne nu beskrive proteinlandskabet i disse undergrupper med hidtil uset detaljegrad og nøjagtighed.
De kan nu sammenligne proteinlandskabet i tumorceller med proteinlandskabet i raskt væv fra samme patient og dermed identificere den proteinubalance, der er årsag til sygdommen, og som potentielt set kan fungere som et effektivt lægemiddelmål.
“Dette har aldrig tidligere være gjort på proteinniveau, men de avancerede faciliteter og forskergrupper ved Novo Nordisk Foundation Center for Protein Research har nu gjort det muligt. Vi har taget det bedste fra to verdener – mikroskopi og proteinforskning – og udviklet en metode, der er hundrede gange mere sensitiv end samtlige tidligere metoder,” siger professor Matthias Mann, Novo Nordisk Foundation Center for Protein Research, Det Sundhedsvidenskabelige Fakultet, Københavns Universitet og Max Planck Institute of Biochemistry.
Det nye studie, “AI-driven Deep Visual Proteomics defines cell identity and heterogeneity”, er udgivet via en såkaldt preprint server med henblik på at gøre det tilgængeligt for offentligheden og andre forskere så hurtigt som muligt. Det er imidlertid stadig under peer review og er således endnu ikke udgivet i et videnskabeligt tidsskrift.
Kontakt:
Lektor Andreas Mund
+45 35 32 50 69
andreas.mund@cpr.ku.dk
Professor Matthias Mann
matthias.mann@cpr.ku.dk
Kommunikationskonsulent Amanda Nybroe Rohde
+45 23 64 94 25
amanda.rohde@sund.ku.dk
